编者注:支持15美元.美国国立卫生研究院颁发的400万奖金, 新罕布什尔州IDeA生物医学研究卓越网络(NH INBRE)旨在提高该州的研究能力和其工作人员的科学知识. 领导联盟的是达特茅斯学院, 负责监督NH INBRE拨款的发放,并为联盟中的合作伙伴提供技术支持和指导, 包括中. 这是一个三部分系列的第二部分,介绍了参与NH INBRE的主要研究教师和学生.
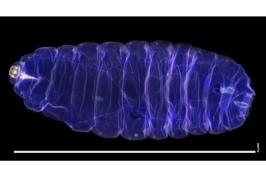
如今,DNA测序不仅仅是遗传学家的专利. 主要研究的Win Watson是一位动物学家,他想更好地了解控制马蹄蟹等动物有节奏活动的内部时钟, 或者叫波吕斐摩斯. 找到构成这种动物生物钟的分子, 了解它的基因组是有帮助的, 或者完整的基因含量.
在过去, 可能是几个月, 科学家可能要经过数年的实验室工作,才能获得沃森等项目中使用的一小部分数据, 但有了下一代测序技术,科学家们现在就可以获得这些数据了, 没有所有的实验室工作.
一种先进的实验室仪器可以对马蹄蟹的DNA进行测序, 把它翻译成四个基本单位,用字母A表示, T, G和C. 然后,沃森和他的合作者使用分析技术——生物信息学——从测序器识别的字母串中收集有意义的遗传信息.
生物信息学已经变得越来越重要,因为生物学家在从进化到生态学等领域使用DNA测序来研究他们周围的世界. 无论你在生物学的哪个领域,生物信息学都是一项核心技能. 凯利·托马斯(Kelley Thomas)领导着NH INBRE在主要研究的生物信息学核心设施.
然而,这是一项相对较新的技能. “DNA测序技术代表了一个非常快速和廉价的真正机会, 但它发生得如此之快,以至于比科学家的职业生涯还要快,托马斯说. “我这个年纪的人在学校里都没有受过这样的训练.”
主要研究核心设施为全州从事NH INBRE项目涉及DNA测序的学校的学生和教师提供生物信息学培训和咨询. (另一个生物信息学核心在达特茅斯大学盖泽尔医学院,由遗传学教授杰森·摩尔指导, 谁在主要研究有一个联合预约.去年,主要研究获得了美国国家科学基金会的资助,为其哈伯德基因组研究中心购买了一台最新的最先进的DNA测序仪. 该测序器支持一系列NH INBRE研究, 从无脊椎动物生物节律与人类健康应用的研究,到大海湾河口微生物群落对污染沉积物的反应研究.
“这些项目是基于我们随后分析的测序数据,冈本和说, 教授生物信息学课程的生物化学研究生. “没有测序数据就没有项目. 当然,这也有一个缺点,因为数据非常庞大. 这就是研讨会的目的. The students come in baffled by these huge sequences of data; they’re almost scared. 然后他们自信地离开. 我很喜欢.”
不到十年前, 通过将DNA片段克隆到细菌中来鉴定一组蛋白质可能需要两到三年的时间. 现在,使用主要研究的Illumina HiSeq 2000,大约需要两周的时间,而且花费更少.
托马斯将这些方法与使用图书馆的卡片目录查找信息和使用谷歌搜索进行比较. 对于后者,“你需要走出很多步骤. 你让事情变得非常直接. Both involve a lot of information; the difference is in how you sort through it.”
今天, 排序过程使用计算机程序来理解可能达到数百gb的大量数据. CLC Genomics Workbench组装短, 将DNA片段重叠成更长的序列来编码基因, Okamoto说. 从那里, 这是一个寻找与其他基因组已经测序的生物体共享的蛋白质的问题. 找到马蹄蟹编码时钟蛋白的基因, 例如, 研究人员寻找与果蝇时钟蛋白已知序列相似的DNA区域. 这相当于在电子文档中搜索一个单词, 尽管果蝇的基因不会和马蹄蟹的基因完全匹配.
冈本说:“这是弄清动物身体特征的垫脚石。.
最初由:
是不是今天
由索尼娅·谢尔(Sonia Scherr)撰写